Spatial organization and functions of Chk1 activation by TopBP1 biomolecular condensates
Tom Egger, Laura Morano, Marie-Pierre Blanchard, Jihane Basbous, Angelos Constantinou
Bases moléculaires de pathologies humaines
Les mécanismes de réparation de l'ADN sont essentiels pour la santé et la survie des organismes. Par conséquent, tous les organismes vivants ont développé divers mécanismes de réparation des lésions de l’ADN. Des défauts héréditaires des mécanismes de réparation de l’ADN sont la cause sous-jacente de maladies susceptibles de provoquer un cancer, comme l'anémie de Fanconi, une maladie caractérisée par une incapacité à réparer les pontages intercaténaires de l’ADN ainsi que les fourches de réplication bloquées. D’autre part, le remaniement métabolique associé à la carcinogenèse induit des lésions de l'ADN. Par conséquent, les cellules cancéreuses dépendent des réponses de la cellule aux lésions de l'ADN pour maintenir leur croissance en présence d'une charge élevée de lésions endogènes ainsi que durant la chimiothérapie.
Notre objectif est de comprendre les mécanismes biochimiques qui sous-tendent les réponses cellulaires aux lésions de l'ADN. Ces mécanismes sont déterminants pour la croissance tumorale et la résistance aux chimiothérapies. Ainsi, nous avons analysé systématiquement la composition protéique des sites de réplication dans des conditions basales et en réponse à une variété d'agents qui induisent des lésions de l’ADN afin d'identifier les protéines qui assurent la progression des fourches de réplication.
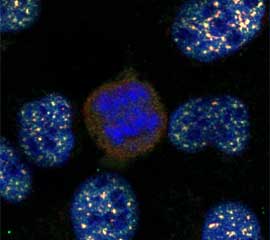
Ces dernières années, nous nous sommes principalement intéressés aux mécanismes et aux fonctions de la compartimentalisation dans la réponse aux lésions de l'ADN. Les protéines impliquées dans la détection, la signalisation et la réparation des lésions de l'ADN s'accumulent généralement dans des sites nucléaires distincts, visualisés par microscopie sous forme de foyers nucléaires. Nous considérons ces foyers nucléaires comme des compartiments sans membrane qui contrôlent l'organisation spatio-temporelle des réponses aux lésions de l'ADN.
Notre modèle de travail est que le recrutement de protéines multivalentes au niveau des lésions de l'ADN nuclée l'assemblage des foyers de réparation. La formation de ces foyers est déclenchée par des modifications post-traductionnelles qui augmentent les interactions entre protéines. Ces interactions coopératives et réversibles favorisent la formation de réseaux protéiques de taille micrométrique qui remplissent les fonctions de compartiments subcellulaires. Par exemple, nous avons récemment découvert que la protéine d’échafaudage TOPBP1 forme des foyers nucléaires qui déclenchent l’activation de la voie de signalisation ATR/Chk1.
Nous combinons des approches de biologie moléculaire, de biochimie, d'optogénétique et de microscopie à haute résolution pour comprendre les mécanismes, l'organisation interne et les fonctions qui découlent spécifiquement de l'assemblage de compartiments sans membrane impliqués dans la réponse des cellules aux lésions de l’ADN.
Tom Egger, Laura Morano, Marie-Pierre Blanchard, Jihane Basbous, Angelos Constantinou
ALGHOUL Emile, BASBOUS Jihane, CONSTANTINOU Angelos
Emile Alghoul, Matteo Paloni, Arato Takedachi, Serge Urbach, Alessandro Barducci, Pierre-Henri Gaillard, Jihane Basbous, Angelos Constantinou
Ovejero S, Viziteu E, Dutrieux L, Devin J, Lin YL, Alaterre E, Jourdan M, Basbous J, Requirand G, Robert N, de Boussac H, Seckinger A, Hose D, Vincent L, Herbaux C, Constantinou A, Pasero P, Moreaux J
+
Modifications épigénétiques et instabilité génomique dans les cellules B normales et tumorales
+ Maintien de l'intégrité du génome au cours de la réplication
Domagoj Vugic, Isaac Dumoulin, Charlotte Martin, Anna Minello, Lucia Alvaro-Aranda, Jesus Gomez-Escudero , Rady Chaaban , Rana Lebdy , Catharina von Nicolai , Virginie Boucherit , Cyril Ribeyre , Angelos Constantinou, Aura Carreira
Alghoul E, Basbous J, Constantinou A
Frattini C, Promonet A, Alghoul E, Vidal-Eychenie S, Lamarque M, Blanchard MP, Urbach S, Basbous J, Constantinou A
Basbous J, Aze A, Chaloin L, Lebdy R, Hodroj D, Ribeyre C, Larroque M, Shepard C, Kim B, Pruvost A, Moreaux J, Maiorano D, Mechali M, Constantinou A
+
Instabilité Génétique et Cancer
Basbous J, Aze A, Chaloin L, Lebdy R, Hodroj D, Ribeyre C, Larroque M, Shepard C, Kim B, Pruvost A, Moreaux J, Maiorano D, Mechali M, Constantinou A
+
Instabilité Génétique et Cancer
Basbous J, Constantinou A
Kumbhar R, Vidal-Eychenié S, Kontopoulos DG, Larroque M, Larroque C, Basbous J, Kossida S, Ribeyre C, Constantinou A
+ IMGT® - le système d'information international en ImMunoGénéTique
Viziteu E, Klein B, Basbous J, Lin YL, Hirtz C, Gourzones C, Tiers L, Bruyer A, Vincent L, Grandmougin C, Seckinger A, Goldschmidt H, Constantinou A, Pasero P, Hose D, Moreaux J.
+
Maintien de l'intégrité du génome au cours de la réplication
Moriel-Carretero, M., Ovejero, S., Gerus-Durand, M., Vryzas, D., Constantinou, A.
Ribeyre C, Zellweger R, Chauvin M, Bec N, Larroque C, Lopes M, Constantinou A
Alsafadi S, Houy A, Battistella A, Popova T, Wassef M, Henry E, Tirode F, Constantinou A, Piperno-Neumann S, Roman-Roman S, Dutertre M, Stern MH
Bret C, Klein B, Cartron G, Schved JF, Constantinou A, Pasero P, Moreaux J.
+ Maintien de l'intégrité du génome au cours de la réplication
Laguette,N., Bregnard, C., Hue, P., Basbous, J., Yatim, A., Larroque, M., Kirchhoff, F., Constantinou, A., Sobhian, B., Benkirane, M.
+
Kassambara A, Gourzones-Dmitriev C, Sahota S, Rème T, Moreaux J, Goldschmidt H, Constantinou A, Pasero P, Hose D, Klein B
+ Maintien de l'intégrité du génome au cours de la réplication
Gourzones-Dmitriev C, Kassambara A, Sahota S, Rème T, Moreaux J, Bourquard P, Hose D, Pasero P, Constantinou A, Klein B.
+
Maintien de l'intégrité du génome au cours de la réplication
Lossaint G, Larroque M, Ribeyre C, Bec N, Larroque C, Décaillet C, Gari K, Constantinou A.
Vidal-Eychenié S, Décaillet C, Basbous J, Constantinou A.
Constantinou, A
Soutenue par Rana Lebdy le 18/03/2022 sous la direction de Cyril Ribeyre
 Rôle de la condensation des protéines en réponse aux dommages de l’ADN
Rôle de la condensation des protéines en réponse aux dommages de l’ADN
Par Jihane Basbous le 02/12/2021
Soutenue par Ramhari Kumbhar le 16/09/2016 sous la direction de Angelos Constantinou et de Cyril Ribeyre, Montpellier
