Mutation hotspots during meiosis
Baudat, F. de Massy, B.
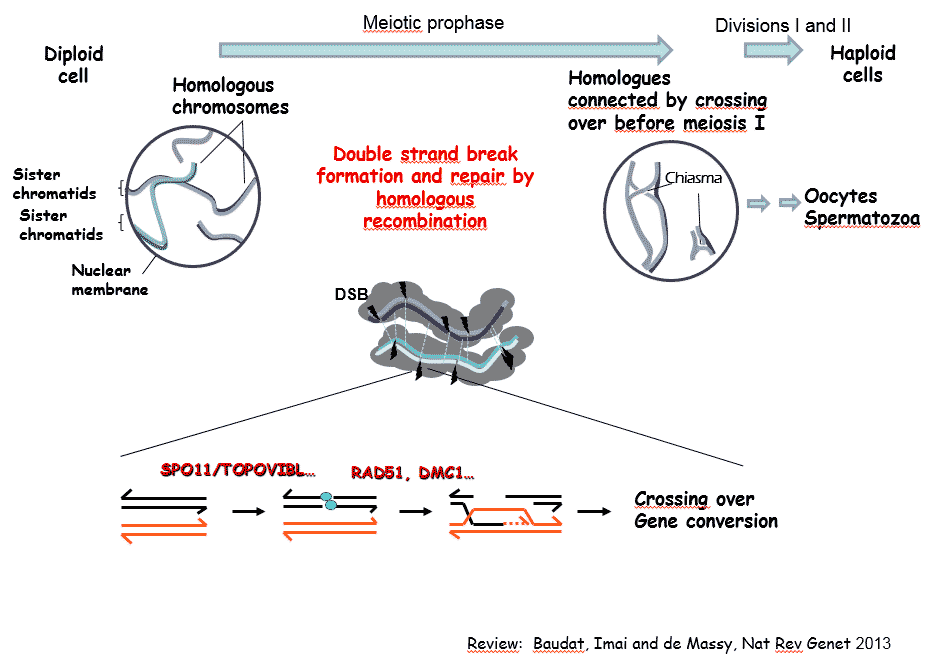
La méiose est une division cellulaire spécialisée qui permet chez les organismes à reproduction sexuée de former des cellules haploïdes à partir de cellules diploïdes. Ceci est réalisé par un cycle cellulaire constitué d’une phase de réplication de l’ADN suivi par deux divisions. La ségrégation réductionnelle des chromosomes lors de la première division de méiose nécessite l’établissement de connections entre chromosomes homologues. Celles-ci sont établies pendant la prophase de la première division par recombinaison homologue qui génère des échanges réciproques, appelés crossing over, entre homologues. L’absence de crossing over conduit le plus souvent à des défauts de ségrégation et à la stérilité. Des altérations du programme de recombinaison peuvent aussi conduire à de l’instabilité génomique et à des aneuploïdies. Par ailleurs, les événements de recombinaison homologues en méiose déterminent les associations génétiques et contribuent à la diversité des génomes et à leur évolution.
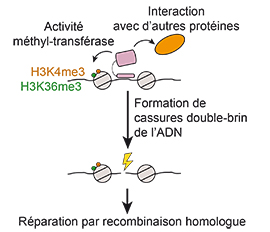
Notre equipe s’intéresse à plusieurs aspects du mécanisme moléculaire de la recombinaison méiotique et de ses implications évolutives en utilisant la souris comme système modèle. La recombinaison méiotique est initiée par l’induction programmée de centaines de cassures double brin de l’ADN dont la réparation conduit à la formation de crossing over et conversion géniques. Les principales étapes et facteurs impliqués dans ces processus ont été conservés au cours de l’évolution.
Baudat, F. de Massy, B.
Laroussi, H. Juarez-Martinez, A. B. Le Roy, A. Boeri Erba, E. Gabel, F. de Massy, B. Kadlec, J.
Nore A., Juarez-Martinez A. B., Clement J., Brun C., Diagouraga B., Laroussi H., Grey C., Bourbon H. M., Kadlec J., Robert T., de Massy B.
Brinkmeier J., Coelho S., de Massy B., Bourbon H. M.
Ihsan Dereli, Marcello Stanzione, Fabrizio Olmeda, Frantzeskos Papanikos, Marek Baumann, Sevgican Demir, Fabrizia Carofiglio, Julian Lange, Bernard de Massy, Willy M Baarends, James Turner, Steffen Rulands, Attila Tóth
Grey C, de Massy B
Zhong-Yi Wang, Evgeny Leushkin, Angélica Liechti, Svetlana Ovchinnikova, Katharina Mößinger, Thoomke Brüning, Coralie Rummel, Frank Grützner, Margarida Cardoso-Moreira, Peggy Janich, David Gatfield, Boubou Diagouraga, Bernard de Massy, Mark E. Gill, Antoine H. F. M. Peters, Simon Anders & Henrik Kaessmann
Imai Y, Biot M, Clément J, Teragaki M, Urbach S, Robert T, Baudat F, Grey C, de Massy B
Biot M, de Massy B
Papanikos F, Clément JAJ, Testa E, Ravindranathan R, Grey C, Dereli I, Bondarieva A, Valerio-Cabrera S, Stanzione M, Schleiffer A, Jansa P, Lustyk D, Fei JF, Adams IR, Forejt J, Barchi M, de Massy B, Toth A
Baudat F, de Massy B, Veyrunes F
Kumar R, Oliver C, Brun C, Juarez-Martinez AB, Tarabay Y, Kadlec J, de Massy B
Grey C, Baudat F, de Massy B
Diagouraga B, Clément JAJ, Duret L, Kadlec J, de Massy B, Baudat F
Clément J, de Massy B
Robert, T, De Massy, B, Grelon, M.
Imai Y, Baudat F, Taillepierre M, Stanzione M, Toth A, de Massy B.
Grey C, Clément JA, Buard J, Leblanc B, Gut I, Gut M, Duret L, de Massy B.
Stanzione M, Baumann M, Papanikos F, Dereli I, Lange J, Ramlal A, Tränkner D, Shibuya H, de Massy B, Watanabe Y, Jasin M, Keeney S, Tóth A
Robert T, Vrielynck N, Mézard C, de Massy B, Grelon M
Robert T, Nore A, Brun C, Maffre C, Crimi B, Bourbon HM, de Massy B
Grey C, Espeut J, Ametsitsi R, Kumar R, Luksza M, Brun C, Verlhac MH, Suja JÁ, de Massy B
Doyen CM1, Chalkley GE1, Voets O1, Bezstarosti K2, Demmers JA2, Moshkin YM1, Verrijzer CP3.
Borde V, de Massy B
Kumar R, Ghyselinck N, Ishiguro KI, Watanabe Y, Kouznetsova A, Höög C, Strong E, Schimenti J, Daniel K, Toth A, de Massy B.
Rahmoun M, Perez J, Saunders PA, Boizet-Bonhoure B, Wilhelm D, Poulat F, Veyrunes F.
+
Développement et Pathologie de la Gonade
de Massy B.
Cole F, Baudat F, Grey C, Keeney S, de Massy B, Jasin M.
Buard J, Rivals E, Dunoyer de Segonzac D, Garres C, Caminade P, de Massy B, Boursot P.
Psomas C, Lavigne JP, Barbuat C, Trabelsi S, Ghosn J, Lascoux-Combe C, Flandre P, Cuzin L, Reynes J, Autran B, Corbeau P.
+
Domiciliation, activation immunitaire et infection
Baudat F, Imai Y, de Massy B.
Wu H, Mathioudakis N, Diagouraga B, Dong A, Dombrovski L, Baudat F, Cusack S, de Massy B, Kadlec J.
De massy, B.
Soumillon M, Necsulea A, Weier M, Brawand D, Zhang X, Gu H, Barthès P, Kokkinaki M, Nef S, Gnirke A, Dym M, de Massy B, Mikkelsen TS, Kaessmann H.
Carofiglio F, Inagaki A, de Vries S, Wassenaar E, Schoenmakers S, Vermeulen C, van Cappellen WA, Sleddens-Linkels E, Grootegoed JA, Te Riele HP, de Massy B, Baarends WM.
Ishmukhametova A, Chen JM, Bernard R, de Massy B, Baudat F, Boyer A, Méchin D, Thorel D, Chabrol B, Vincent MC, Van Kien PK, Claustres M, Tuffery-Giraud S.
Orsi GA, Algazeery A, Meyer RE, Capri M, Sapey-Triomphe LM, Horard B, Gruffat H, Couble P, Aït-Ahmed O, Loppin B.
Reynolds A, Qiao H, Yang Y, Chen JK, Jackson N, Biswas K, Holloway JK, Baudat F, de Massy B, Wang J, Höög C, Cohen PE, Hunter N.
Borde V, de Massy B.
De Massy, B.
Lutzmann M, Grey C, Traver S, Ganier O, Maya-Mendoza A, Ranisavljevic N, Bernex F, Nishiyama A, Montel N, Gavois E, Forichon L, de Massy B, Méchali M.
+
Réplication et Dynamique du Génome
Romero Y, Vuandaba M, Suarez P, Grey C, Calvel P, Conne B, Pearce D, de Massy B, Hummler E, Nef S.
Flachs P, Mihola O, Simeček P, Gregorová S, Schimenti JC, Matsui Y, Baudat F, de Massy B, Piálek J, Forejt J, Trachtulec Z.
Baudat F, Buard J, Grey C, de Massy B.
Grey C, Barthès P, Chauveau-Le Friec G, Langa F, Baudat F, de Massy B.
Romero Y, Meikar O, Papaioannou MD, Conne B, Grey C, Weier M, Pralong F, De Massy B, Kaessmann H, Vassalli JD, Kotaja N, Nef S.
Lichten, M., de Massy, B.
Grey C, Sommermeyer V, Borde V, de Massy B
Grad I, Cederroth CR, Walicki J, Grey C, Barluenga S, Winssinger N, De Massy B, Nef S, Picard D
Meyer, RE., Delaage, M., Rosset, R., Capri, M., Ait-Ahmed, O.
Baudat F, Buard J, Grey C, de Massy B.
Kumar, R., Bourbon, H.M., De Massy, B.
Baudat, F., Buard, J., Grey, C., Fledel-Alon, A., Ober, C., Przeworski, M., Coop, G., de Massy, B.
Buard, J., Barthès, P., Grey, C. and de Massy, B_.
Bolcun-Filas, E., Speed, R., Taggart, M., Grey, C., de Massy, B., Benavente, R., Cooke, HJ.
Grey C, Baudat F, de Massy B.
Baudat, F. and de Massy, B.
Svetlanov A, Baudat F, Cohen PE, de Massy B.
Le Mée, G., Ezzeddine, N., Capri, M., Aït-Ahmed, O.
Baudat, F., de Massy, B.
Baudat, F., de Massy, B
Chicheportiche A, Bernardino-Sgherri J, de Massy B, Dutrillaux B.
Buard, J., De Massy, B.
Bérard S., Buard J. et E. Rivals.
Cosson, B., Gautier-Courteille, C., Maniey, D., Ait-Ahmed, O., Lesimple, M., Osborne, H.B., Paillard, L.
Guettier, C., Sebagh, M., Buard, J., Feneux, D., Ortin-Serrano, M., Gigou, M., Tricottet, V., Reynes, M., Samuel, D., Feray, C.
Alkan, C., Tuzun, E., Buard, J., Lethiec, F., Eichler, EE., Bailey JA., Sahinalp, SC.
Guillon, H., Baudat, F., Grey, C., Liskay, R.M. and de Massy, B
Lemaitre, JM., Bocquet, S., Terret, ME., Namdar, M., Ait-Ahmed, O., Kearsey, S., Verlhac, MH., and Méchali, M.
+
Réplication et Dynamique du Génome
Delaunay J, Le Mée G, Ezzeddine N, Labesse G, Terzian C, Capri M, Aït-Ahmed O.
de Massy, B.
Laurent, A. M., Li, M., Sherman, S., Roizes, G., Buard, J.,
Guillon H. and de Massy B.
Buard J., Brenner C., Jeffreys A.J.
Lopes J., Debrauwere H., Buard J., Nicolas A.
Ezzeddine, N., Paillard, L., Capri, M., Maniey, D., Bassez, T., Aït-Ahmed, O. and Osborne, H.B.
Stead JD, Buard J, Todd JA, Jeffreys AJ.
Debrauwere H., Nicolas A., Vergnaud G., Buard J., Tessier J. et Aubert D.
Buard, J., Shone, A.C. and Jeffreys, A.J.
Buard J., Collick A., Brown J. and Jeffreys, A.J.
Metzler-Guillemain, C. et de Massy B.
Debrauwere H, Buard J, Tessier J, Aubert D, Vergnaud G, Nicolas A.
Gangloff, S., De Massy, B., Lane, A., Rothstein, R. and Fabre, F.
Santoni, M.J., Aït-Ahmed, O. and Marilley, M.
Dardhalon, M., De Massy, B., Nicolas, A. and Averbeck, D.
Capri, M., Santoni, M.J., Thomas-Delaage, M. and Aït-Ahmed, O.
Soutenue par Mathilde Biot le 11/03/2022 sous la direction de Bernard De Massy
 HDR “Control of the initiation of meiotic recombination in mouse”
HDR “Control of the initiation of meiotic recombination in mouse”
Par Corinne Grey le 26/05/2021
Soutenue par Julia Brinkmeier le 30-03-2020
Soutenue par Alexandre Nore le 29-11-2018
Soutenue par Denis Dunoyer de Segonzac le 25-11-2016
Soutenue par Boubou Diagouraga le 15-12-2015
Soutenue par Yukiko Imai le 11/12/2015
