Epigenetic Chromatin Regulation
Génétique, biologie cellulaire et développement

La répression transcriptionnelle médiée par des petits ARN non codants est un processus fondamental observé chez de nombreux eucaryotes, comme les champignons, les plantes, les mouches, les vers ou les mammifères. L'un de leurs rôles connus est de neutraliser l'activité des éléments transposables (TE), qui sinon peuvent déstabiliser leur génome-hôte et causer diverses maladies. Les petits ARN utilisent leur complémentarité de base avec les TE pour les éteindre spécifiquement. Cependant, la façon dont les cellules assurent la répression de ces derniers sans déranger l’expressions d’autres gènes est encore relativement incomprise.
Le cillié Tetrahymena identifie les séquences dérivées de TE par un mécanisme de comparaison des génomes germinal et somatique, médié par de petits ARN lors d’une élimination programmée de l'ADN, fournissant des exemples fascinants de régulation épigénetique du génome, ainsi que d’importants renseignements sur l'interaction entre TE et génomes-hôtes. Parce que l'élimination programmée de l'ADN peut être induite de façon synchrone et à grande échelle en laboratoire, elle se trouve être un modèle très utile à l'étude génétique et biochimique de la régulation de la chromatine par petits ARN.
À l'aide de ce protozoaire, nous cherchons à comprendre comment les cellules accumulent de façon spécifique de petits ARN à partir de séquences apparentées aux TE; comment les cellules utilisent ces petits ARN pour identifier ces séquences de type TE; et comment une voie de petits ARN établit un environnement de chromatine inhibée (hétérochromatine) sur des séquences apparentées aux TE.
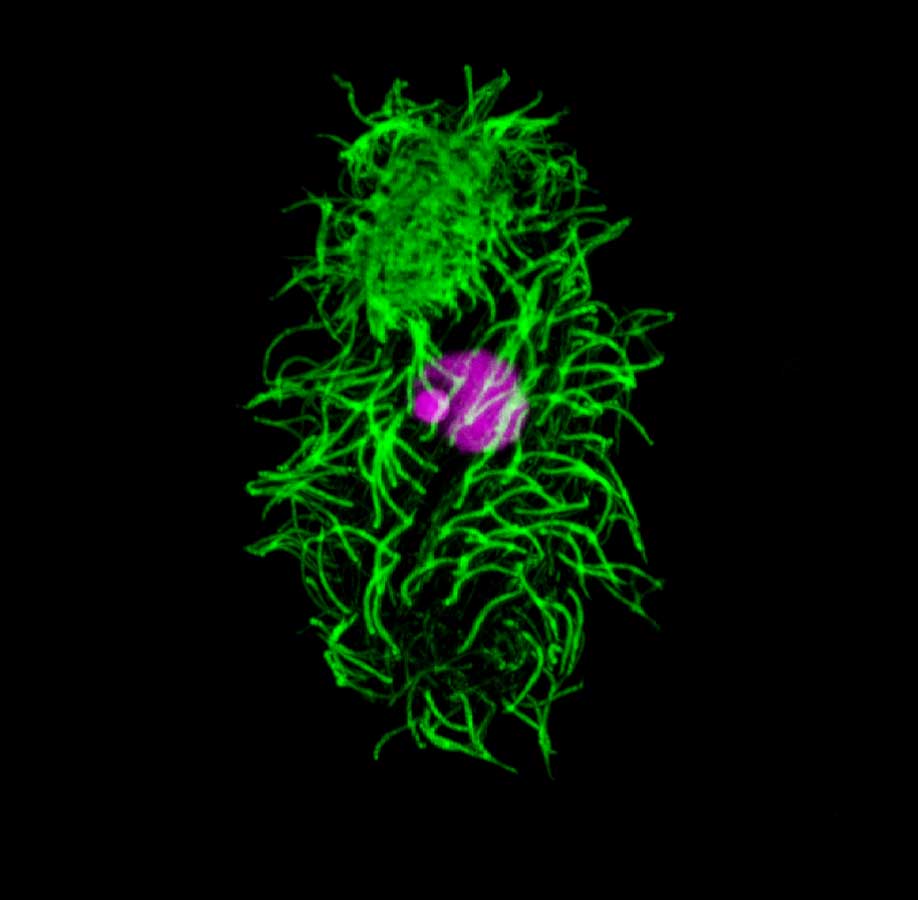
Tetrahymena thermophila. La Tétrahyména est un eucaryote unicellulaire. Elle possède de nombreux cils sur sa surface cellulaire (vert = coloration anti-alpha tubuline), deux noyaux différents (violet), le plus petit les micronoyaux de la lignée germinale (MIC) et le plus grand le macronoyau somatique (MAC).
Publications de l'équipe
Programmed DNA elimination: New metazoan models.
Mochizuki K
PIWI-Directed DNA Elimination for Tetrahymena Genetics.
Shehzada S, Mochizuki K
Arrested crossover precursor structures form stable homologous bonds in a Tetrahymena meiotic mutant.
Tian M, Mochizuki K, Loidl J
Genomes: Programmed DNA Elimination in a Parasitic Nematode.
Mochizuki K
Non-coding RNA Transcription in Tetrahymena Meiotic Nuclei Requires Dedicated Mediator Complex-Associated Proteins.
Tian M, Mochizuki K, Loidl J
Diversification of small RNA amplification mechanisms for targeting transposon-related sequences in ciliates.
Mutazono M, Noto T, Mochizuki K.
Small RNA-Mediated trans-Nuclear and trans-Element Communications in Tetrahymena DNA Elimination.
Noto T, Mochizuki K
Whats, hows and whys of programmed DNA elimination in Tetrahymena.
Noto T, Mochizuki K
A Zip3-like protein plays a role in crossover formation in the SC-less meiosis of the protist Tetrahymena.
Shodhan A, Kataoka K, Mochizuki K, Novatchkova M, Loidl J
Negative Regulators of an RNAi-Heterochromatin Positive Feedback Loop Safeguard Somatic Genome Integrity in Tetrahymena
Suhren JH, Noto T, Kataoka K, Gao S, Liu Y, Mochizuki K.
Heterochromatin aggregation during DNA elimination in Tetrahymena is facilitated by a prion-like protein.
Kataoka K, Mochizuki K
Structure of the germline genome of Tetrahymena thermophila and relationship to the massively rearranged somatic genome.
Hamilton EP, Kapusta A, Huvos PE, Bidwell SL, Zafar N, Tang H, Hadjithomas M, Krishnakumar V, Badger JH, Caler EV, Russ C, Zeng Q, Fan L, Levin JZ, Shea T, Young SK, Hegarty R, Daza R, Gujja S, Wortman JR, Birren BW, Nusbaum C, Thomas J, Carey CM, Pritham EJ, Feschotte C, Noto T, Mochizuki K, Papazyan R, Taverna SD, Dear PH, Cassidy-Hanley DM, Xiong J, Miao W, Orias E, Coyne RS
Phosphorylation of an HP1-like protein is a prerequisite for heterochromatin body formation in Tetrahymena DNA elimination.
Kataoka K, Noto T, Mochizuki K
Phosphorylation of an HP1-like Protein Regulates Heterochromatin Body Assembly for DNA Elimination.
Kataoka K, Mochizuki K
Targeted Gene Disruption by Ectopic Induction of DNA Elimination in Tetrahymena.
Hayashi A, Mochizuki K
Small-RNA-Mediated Genome-wide trans-Recognition Network in Tetrahymena DNA Elimination.
Noto T, Kataoka K, Suhren JH, Hayashi A, Woolcock KJ, Gorovsky MA, Mochizuki K
A Tetrahymena Hsp90 co-chaperone promotes siRNA loading by ATP-dependent and ATP-independent mechanisms.
Woehrer SL, Aronica L, Suhren JH, Busch CJ, Noto T, Mochizuki K
The taming of the shrew: Regulation of a catalytically active domesticated transposase.
Vogt A, Mochizuki K
A domesticated PiggyBac transposase interacts with heterochromatin and catalyzes reproducible DNA elimination in Tetrahymena.
Vogt A, Mochizuki K
Epigenetics of ciliates.
Chalker DL, Meyer E, Mochizuki K
Analysis of Piwi-loaded small RNAs in Tetrahymena.
Noto T, Kurth HM, Mochizuki K
Transposon domestication versus mutualism in ciliate genome rearrangements.
Vogt A, Goldman AD, Mochizuki K, Landweber LF
Biased transcription and selective degradation of small RNAs shape the pattern of DNA elimination in Tetrahymena.
Schoeberl UE, Kurth HM, Noto T, Mochizuki K
Developmentally programmed, RNA-directed genome rearrangement in Tetrahymena.
Mochizuki K
DNA rearrangements directed by non-coding RNAs in ciliates.
Mochizuki K
Programmed DNA elimination in Tetrahymena: a small RNA-mediated genome surveillance mechanism.
Kataoka K, Mochizuki K
Keeping the soma free of transposons: programmed DNA elimination in ciliates.
Schoeberl UE, Mochizuki K
RNA-directed epigenetic regulation of DNA rearrangements.
Mochizuki K
Modules for C-terminal epitope tagging of Tetrahymena genes.
Kataoka K, Schoeberl UE, Mochizuki K
MRE11 and COM1/SAE2 are required for double-strand break repair and efficient chromosome pairing during meiosis of the protist Tetrahymena.
Lukaszewicz A, Howard-Till RA, Novatchkova M, Mochizuki K, Loidl J
A domesticated piggyBac transposase plays key roles in heterochromatin dynamics and DNA cleavage during programmed DNA deletion in Tetrahymena thermophila.
Cheng CY, Vogt A, Mochizuki K, Yao MC
The Tetrahymena argonaute-binding protein Giw1p directs a mature argonaute-siRNA complex to the nucleus.
Noto T, Kurth HM, Kataoka K, Aronica L, DeSouza LV, Siu KW, Pearlman RE, Gorovsky MA, Mochizuki K
Two GW repeat proteins interact with Tetrahymena thermophila argonaute and promote genome rearrangement.
Bednenko J, Noto T, DeSouza LV, Siu KW, Pearlman RE, Mochizuki K, Gorovsky MA
Non-coding RNA: a bridge between small RNA and DNA.
Kurth HM, Mochizuki K
2'-O-methylation stabilizes Piwi-associated small RNAs and ensures DNA elimination in Tetrahymena.
Kurth HM, Mochizuki K
High efficiency transformation of Tetrahymena using a codon-optimized neomycin resistance gene.
Mochizuki K
Study of an RNA helicase implicates small RNA-noncoding RNA interactions in programmed DNA elimination in Tetrahymena.
Aronica L, Bednenko J, Noto T, DeSouza LV, Siu KW, Loidl J, Pearlman RE, Gorovsky MA, Mochizuki K
DNA double-strand breaks, but not crossovers, are required for the reorganization of meiotic nuclei in Tetrahymena.
Mochizuki K, Novatchkova M, Loidl J
A Dicer-like protein in Tetrahymena has distinct functions in genome rearrangement, chromosome segregation, and meiotic prophase.
Mochizuki K, Gorovsky MA
RNA polymerase II localizes in Tetrahymena thermophila meiotic micronuclei when micronuclear transcription associated with genome rearrangement occurs.
Mochizuki K, Gorovsky MA
Conjugation-specific small RNAs in Tetrahymena have predicted properties of scan (scn) RNAs involved in genome rearrangement.
Mochizuki K, Gorovsky MA
Histone H3 lysine 9 methylation is required for DNA elimination in developing macronuclei in Tetrahymena.
Liu Y, Mochizuki K, Gorovsky MA
Analysis of a piwi-related gene implicates small RNAs in genome rearrangement in tetrahymena.
Mochizuki K, Fine NA, Fujisawa T, Gorovsky MA
Universal occurrence of the vasa-related genes among metazoans and their germline expression in Hydra.
Mochizuki K, Nishimiya-Fujisawa C, Fujisawa T
Expression and evolutionary conservation of nanos-related genes in Hydra.
Mochizuki K, Sano H, Kobayashi S, Nishimiya-Fujisawa C, Fujisawa T
Thèses et hdr
Régulations du renouvellement des petits ARN lors de l'élimination programmée de l'ADN chez Tetrahymena 28/10/2022
Soutenue par Salman Shehzada sous la direction de Kazufumi Mochizuki - Soutenue le 28-10-2022
Caractérisation des protéines requises dans la formation d’hétérochromatine ARN-médiée 04/12/2020
Soutenue par Elliot Geraud le 04/12/2020