A new perspective on microRNA-guided gene regulation specificity, and its potential generalization to transcription factors and RNA-binding proteins.
Seitz H
Génétique, biologie cellulaire et développement
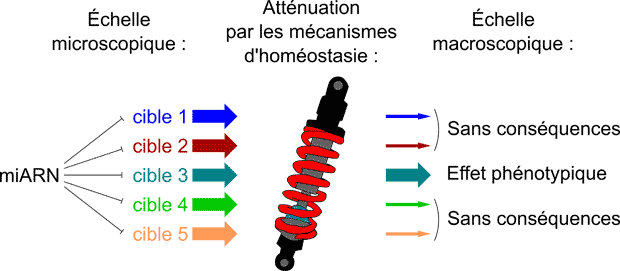
Les microARN (« miARN ») sont de petits régulateurs post-transcriptionnels. La fonction de ces petits ARN chez les Animaux a été bien caractérisée au niveau moléculaire, mais leur rôle est moins bien connu à l'échelle macroscopique : comment les miARN pourraient-ils avoir une fonction biologique, s'ils répriment la plupart de leurs cibles d'un facteur inférieur à 2 (alors que les fluctuations d'expression des gènes entre individus excèdent typiquement un facteur 2, et qu'elles sont tamponnées par les mécanismes d'homéostasie) ?
D'après le dogme actuel, chaque miARN régule des dizaines ou des centaines de cibles, mais plusieurs observations suggèrent que les miARN ont un impact beaucoup plus modeste sur la biologie animale. Nos travaux récents suggèrent également qu'à la fois les méthodes expérimentales et bio-informatiques pour l'identification des cibles de miARN sont lourdement contaminées par des faux positifs : ces faux positifs peuvent être réellement réprimés par les miARN à l'échelle moléculaire, mais une répression aussi faible ne parvient pas à déclencher un phénotype macroscopique pour la plupart des gènes.
Notre travail suggère donc que le rôle biologique des miARN a été largement surestimé. Nous explorons à présent les conséquences pratiques de ce nouveau cadre théorique, en mesurant la contribution d'interactions individuelles entre miARN et cibles, sur des phénotypes globaux in vivo.
Plus généralement, nous proposons une nouvelle vision de la régulation des gènes : une cible régulatrice n'est pas simplement un gène qui est affecté par une voie régulatrice ; c'est un gène qui est affecté suffisamment par la voie régulatrice – l'amplitude de la régulation mesurée doit être confrontée à la robustesse des systèmes biologiques face aux fluctuations.
Contacter le chef d'équipe (Hervé Seitz) par email : herve.seitz[at]igh.cnrs.fr
Seitz H
Busseau I, Mockly S, Houbron É, Somaï H, Seitz H
Houbron É, Mockly S, Rafasse S, Gros N, Muriaux D, Seitz H
Mockly S, Seitz H
Sophie Mockly, Élisabeth Houbron, Hervé Seitz
Hebras J, Marty V, Personnaz J, Mercier P, Krogh N, Nielsen H, Aguirrebengoa M, Seitz H, Pradere JP, Guiard BP, Cavaille J
Patricia Richard, Shuang Feng, Yueh-Lin Tsai, Wencheng Li, Paola Rinchetti, Ubayed Muhith, Juan Irizarry-Cole, Katharine Stolz, Lionel A Sanz, Stella Hartono, Mainul Hoque, Saba Tadesse, Hervé Seitz, Francesco Lotti, Michio Hirano, Frédéric Chédin, Bin Tian, James L Manley
Canzler S, Schor J, Busch W, Schubert K, Rolle-Kampczyk UE, Seitz H, Kamp H, von Bergen M, Buesen R, Hackermüller J
+
Impact systémique des petits ARN régulateurs
Canzler S, Schor J, Busch W, Schubert K, Rolle-Kampczyk UE, Seitz H, Kamp H, von Bergen M, Buesen R, Hackermüller J
+
Impact systémique des petits ARN régulateurs
Mockly S, Seitz H
Amar L, Seitz H
Seitz H
Pinzón N, Bertrand S, Subirana L, Busseau I, Escrivá H, Seitz H
Marlétaz F, Firbas PN, Maeso I, Tena JJ, Bogdanovic O, Perry M, Wyatt CDR, de la Calle-Mustienes E, Bertrand S, Burguera D, Acemel RD, van Heeringen SJ, Naranjo S, Herrera-Ubeda C, Skvortsova K, Jimenez-Gancedo S, Aldea D, Marquez Y, Buono L, Kozmikova I, Permanyer J, Louis A, Albuixech-Crespo B, Le Petillon Y, Leon A, Subirana L, Balwierz PJ, Duckett PE, Farahani E, Aury JM, Mangenot S, Wincker P, Albalat R, Benito-Gutiérrez È, Cañestro C, Castro F, D'Aniello S, Ferrier DEK, Huang S, Laudet V, Marais GAB, Pontarotti P, Schubert M, Seitz H, Somorjai I, Takahashi T, Mirabeau O, Xu A, Yu JK, Carninci P, Martinez-Morales JR, Crollius HR, Kozmik Z, Weirauch MT, Garcia-Fernàndez J, Lister R, Lenhard B, Holland PWH, Escriva H, Gómez-Skarmeta JL, Irimia M
Buesen R, Chorley BN, da Silva Lima B, Daston G, Deferme L, Ebbels T, Gant TW, Goetz A, Greally J, Gribaldo L, Hackermüller J, Hubesch B, Jennen D, Johnson K, Kanno J, Kauffmann HM, Laffont M, McMullen P, Meehan R, Pemberton M, Perdichizzi S, Piersma AH, Sauer UG, Schmidt K, Seitz H, Sumida K, Tollefsen KE, Tong W, Tralau T, van Ravenzwaay B, Weber RJM, Worth A, Yauk C, Poole A
Rodríguez-Martínez, M., Pinzón, N., Ghommidh, C., Beyne, E., Seitz, H., Cayrou, C., Méchali, M.
+
Réplication et Dynamique du Génome
Seitz H
Pinzon, N., Li, B., Martinez, L., Sergeeva, A., Presumey, J., Apparailly, F., Seitz, H
Eckenfelder A, Ségéral E, Pinzón N, Ulveling D, Amadori C, Charpentier M, Nidelet S, Concordet JP, Zagury JF, Paillart JC, Berlioz-Torrent C, Seitz H, Emiliani S, Gallois-Montbrun S.
Aigner A, Buesen R, Gant T, Gooderham N, Greim H, Hackermüller J, Hubesch B, Laffont M, Marczylo E, Meister G, Petrick JS, Rasoulpour RJ, Sauer UG, Schmidt K, Seitz H, Slack F, Sukata T, van der Vies SM, Verhaert J, Witwer KW, Poole A
Hoffmann S, Clauss S, Berger IM, Weiß B, Montalbano A, Röth R, Bucher M, Klier I, Wakili R, Seitz H, Schulze-Bahr E, Katus HA, Flachsbart F, Nebel A, Guenther SP, Bagaev E, Rottbauer W, Kääb S, Just S, Rappold GA.
Royo H, Seitz H, ElInati E, Peters AH, Stadler MB, Turner JM
Tarver JE, Cormier A, Pinzón N, Taylor RS, Carré W, Strittmatter M, Seitz H, Coelho SM, Cock JM
Chambeyron, S., Seitz, H.
+
ARN non codants, épigénétique et stabilité génomique
Moran, Y., Fredman, D., Praher, D., Li Z. L., Meng Wee,L., Rentzsch, F., Zamore,P.D., Technau, U., Seitz H.
Sergeeva, A., Restrepo, N.P., and Seitz, H.
Seitz H, Tushir JS, Zamore PD
Tsutsumi A, Kawamata T, Izumi N, Seitz H, Tomari Y
Soutenue par Sophie Mockly le 08/12/2021 sous la direction de Hervé Seitz
Site web de l’événement : https://internat21.sciencesconf.org/
Site web de l’événement : https://internat.sciencesconf.org
Contenu, en langue française, des cours introductifs donnés pendant la première demi-journée de l’école thématique (introduction sur le RNAi, sur les microARN, et sur la biochimie du complexe RISC ; exercices d’application) : en téléchargement libre ici.
| Collaborateur | But de la collaboration | Publications communes |
|---|---|---|
| Dr. Yukihide TOMARI (université de Tõkyõ) Site web |
Analyse des déterminants structuraux de la biogenèse des microARN | Kawamata et al. (2009) Pubmed Tsutsumi et al. (2011) Pubmed |
| Dr. Ulrich TECHNAU (université de Vienne) Site web |
Analyse fonctionnelle des petits ARN régulateurs chez Nematostella vectensis | Moran et al. (2014) Pubmed |
| Dr. Fabian RENTZSCH (SARS Center) Site web |
Analyse fonctionnelle des petits ARN régulateurs chez Nematostella vectensis | Moran et al. (2014) Pubmed |
| Prof. Phillip D. ZAMORE (école de médecine de l'université du Massachusetts) Site web |
Analyse fonctionnelle des petits ARN régulateurs chez Nematostella vectensis | Moran et al. (2014) Pubmed |
| Dr. James TURNER (Crick Institute, Londres) Site web |
Analyse de l'expression des microARN soumis à un contrôle épigénétique chez les Mammifères | Royo et al. (2015) Pubmed |
| Dr. Denis TAGU (INRA Rennes) Site web |
Caractérisation des petits ARN régulateurs chez le Puceron | |
| Dr. Florence APPARAILLY (IRMB, Montpellier) Site web |
Mesure de la fluctuation inter-individu de l'expression génétique dans les neutrophiles | Pinzón et al. (2017) Pubmed |
| Dr. Marcel MÉCHALI (IGH, Montpellier) Site web |
Identification des origines de réplication du développement précoce du Nématode et caractérisation épigénétique | Rodríguez-Martínez et al. (2017) Pubmed |
| Dr. Mark COCK (UMR 8227, Roscoff) Site web |
Identification des microARN de l'Algue brune Ectocarpus silicosus | Tarver et al. (2015) Pubmed |
| Prof. Gudrun RAPPOLD (UniversitätKlinikum, Heidelberg) Site web |
Identification d'un site de reconnaissance de microARN spécifique de patients atteints de fibrillation atriale | Hoffmann et al. (2016) Pubmed |
| Dr. Hector ESCRIVA (Observatoire océanologique de Banyuls) Site web |
Caractérisation des petits ARN chez le Céphalochordé Branchiostoma lanceolatum |
Marlétaz et al. (2018) Pubmed Pinzón et al. (2019) Pubmed |
| Dr. Sarah GALLOIS-MONTBRUN (Institut Cochin, Paris) Site web |
Identification des sites d'interaction d'Ago2 sur l'ARN viral VIH-1 | Eckenfelder et al. (2017) Pubmed |
| Dr. James Manley (Columbia university, New York, États-Unis) Site web |
Caractérisation des conséquences moléculaires de la répression de SETX dans des cellules humaines | Richard et al. (2020) Pubmed |
| Dr. Jérôme Cavaillé (CBI, Toulouse) Site web |
Évaluation des conséquences moléculaires et physiologiques de la délétion de l’ARN SNORD115 | Hebras et al. (2020) Pubmed |
Scripts et données pour la préparation des figures.
Scripts et données pour la préparation des figures et figures supplémentaires - 56 Ko
Scripts et données pour la préparation des figures et figures supplémentaires - 7,2 Go
Scripts et données intermédiaires
Scripts et données intermédiaires
Scripts et données intermédiaires
Scripts et données pour la préparation de la Table 1
Scripts et données intermédiaires
Scripts et données pour la préparation des 6 figures :
Scripts et données pour la génération des figures 2D, 3B et 4A
Script de conversion (usage : ./read_FCS.py input.fcs output.txt) : script python.
1. Apprentissage d'Unix
2. Les statistiques en biologie moléculaire
3. Apprentissage de R
Contexte :
Les chercheurs CNRS doivent soumettre chaque année un compte-rendu annuel d’activité, et tous les 5 ans, un « rapport d’activité par vague » plus détaillé (avec, au milieu de cette période de 5 ans, un « rapport d’activité à mi-vague »). Suite à la fusion des régions Midi-Pyrénées et Languedoc-Roussillon, les laboratoires de Languedoc-Roussillon ont rejoint la vague des laboratoires de Midi-Pyrénées, repoussant d’une année l’évaluation par vague prévue en 2019 (elle aura lieu en 2020).
D’autre part, les laboratoires sont évalués tous les 5 à 6 ans par un jury international, rassemblé par l’HCERES (anciennement : AERES), qui publie ensuite un compte-rendu d’évaluation (en anglais).
Rapports d’activité et comptes-rendus d’évaluation de notre équipe :
Rapport d'activité à mi-vague 2022 d'Hervé Seitz (décembre 2019 – juin 2022) (en français)
Compte-rendu d’évaluation 2020 de notre équipe par l’HCERES (en anglais)
Rapport d'activité par vague 2020 d'Hervé Seitz (janvier 2014 – décembre 2019) (en français)
Rapport d’activité à mi-vague 2016 d’Hervé Seitz (janvier 2014 – septembre 2016) (en français)
Compte-rendu d’évaluation 2014 de notre équipe par l’AERES (en anglais)
Rapport d’activité par vague 2014 d’Hervé Seitz (janvier 2009 – décembre 2013) (en français)
« La technologie CRISPR/Cas9 »
(compte-rendu de la conférence donnée par Hervé Seitz au colloque « Quelles limites pour les technosciences en santé ? » à Clermont-Ferrand le 13 mars 2018, et publié dans le n°15 de la Revue du Centre Michel de l'Hospital).
Parallèle entre l'informatique et la génétique
(édition du génome, débuggage, matérialité de l'information, ...) (interview donnée dans le cadre des Tic-Talks du LIG).
Les statistiques en science expérimentale. Principe, limitations, erreurs courantes, illustrées par les rumeurs pseudo-scientifiques sur la Covid-19
(visio-conférence donnée le 13 mars 2021 sur invitation du cercle zététique du Languedoc-Roussillon ; diaporama cliquable accessible ici).
